运行环境推荐
anaconda3,集成了常用的用于科学计算的包,以及python解释器(本例使用的解释器版本为3.11.5)
编辑器或者IDE:Vscode或者pycharm
程序实现 摘要 本文将SGA遗传算法按照ppt的编写分为八个模块 (python文件)进行阐述
分别是:
geneEncoding.py
ga.py(主功能模块,运行此模块即可运行遗传算法)
best.py
calfitValue.py
calobjValue.py
crossover.py
mutation.py
selection.py
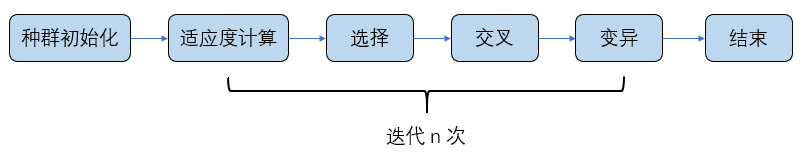
具体的流程图解释如下:
种群初始化:对参数进行编码,适应度函数的设计,以及初始群体的设定
适应度计算:为每次群体中的个体赋予适应度,方便后面遗传操作的进行
选择操作:对群体进行选择 ,选择出适应度值较大的一部分优势群体;
交叉操作:对优势种群进行 “交配 ”,更容易产生优秀的个体;
变异操作:对染色体中的基因用一定的变异方法进行变异
下面以计算函数最大值为例,阐述SGA的具体步骤
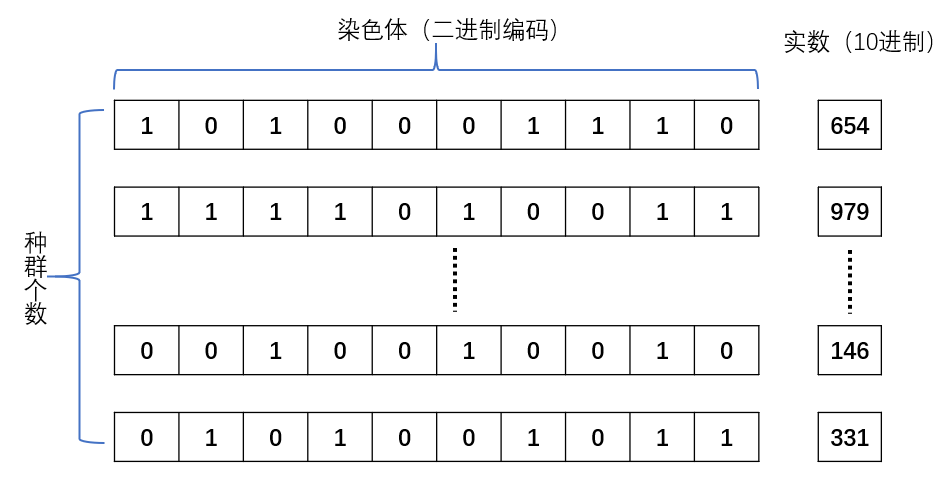
基因编码 在本例中,染色体的编码方式采用二进制编码 ,即用若干二进制数表示一个个体,将原问题的解空间映射到位串空间B={0,1}上
相应模块geneEncoding.py如下
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 import random""" @brief 生成一个二元组的种群,其中种群内的个体以二进制的形式来表示 @param pop_size 整个种群的个体数量 chrom_length 染色体的长度,其实就是二元组内元素的二进制数的个数, 比如当chrom_length=4,种群内的一个个体可以表示为[1,1,0,1] @retval 返回一个种群,由于初始化时第一个元素是空列表[],因此返回的是pop[1:] """ def geneEncoding (pop_size, chrom_length ): pop = [[]] for i in range (pop_size): temp = [] for j in range (chrom_length): temp.append(random.randint(0 , 1 )) pop.append(temp) return pop[1 :] if __name__ == "__main__" : pop_size = 5 chrom_length = 2 pop = geneEncoding(pop_size, chrom_length) print (pop, "\n" ) print (len (pop), "\n" )
可以注意到,经过编码生成后的种群,可以看作是一个二维矩阵,其中矩阵的size=(种群大小,染色体编码长度)
个体适应度的计算 适应度函数得出的值越大表明个体越优秀,所以一般情况下,在求解函数最大值的时候,适应度函数就是求解函数本身 ,求解最小值的时候适应度函数就是函数的倒数。在本例中求取最大值,所以适应度函数就是函数本身 。
相应模块calobjValue.py内容如下
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 import math""" @brief 解码,将传进来的二进制序列转换成十进制 @param pop_size 种群的个体数量 chrom_length 染色体的长度 @retval 十进制序列 """ def decodechrom (pop, chrom_length ): temp = [] for i in range (len (pop)): t = 0 for j in range (chrom_length): t += pop[i][j] * (math.pow (2 , j)) temp.append(t) return temp """ @brief 根据适应度函数,计算种群中个体的适应度 @param pop 种群,一个二元组 chrom_length 染色体的长度 max_value 染色体长度允许的最大值 @retval 个体适应度的列表 """ def calobjValue (pop, chrom_length, max_value ): temp1 = [] obj_value = [] temp1 = decodechrom(pop, chrom_length) for i in range (len (temp1)): x = temp1[i] * max_value / (math.pow (2 , chrom_length) - 1 ) obj_value.append(10 * math.sin(5 * x) + 7 * math.cos(4 * x)) return obj_value if __name__ == "__main__" : pop_size = 5 pop = [[0 , 0 , 1 ], [0 , 1 , 0 ], [1 , 0 , 0 ]] test = calobjValue(pop, 3 , 10 ) print (test)
淘汰 在上一步根据适应度函数计算出个体相应的适应度之后,得出来的结果中,有些个体对应的适应度会是负值
说明这些个体,是不适合**“生存”**的,我们需要将这部分适应度为负值的个体进行淘汰和去除
具体方法如calfitValue.py所示:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 """ @brief 对种群中不适合的个体进行淘汰,其实就是去除负值 @param obj_value 个体适应度的列表 @retval 个体适应度淘汰后的列表 """ def calfitValue (obj_value ): fit_value = [] c_min = 0 for i in range (len (obj_value)): if obj_value[i] + c_min > 0 : temp = c_min + obj_value[i] else : temp = 0.0 fit_value.append(temp) return fit_value if __name__ == "__main__" : pass
保存最优解 在上一步去除掉本次迭代中的不适应个体后,我们需要从当前种群中保存本次迭代中的最优个体以及对应的基因 ,这里采取的方法是直接从适应度列表中找一个适应度最大 的个体
具体实现如模块best.py
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 """ @brief 找出最优解和最优解的基因编码 @param pop 种群,一个二元组 fit_value 适应度列表 @retval 最优解及其基因 """ def best (pop, fit_value ): best_individual = [] best_fit = fit_value[0 ] for i in range (1 , len (pop)): if fit_value[i] > best_fit: best_fit = fit_value[i] best_individual = pop[i] return [best_individual, best_fit] if __name__ == "__main__" : pass
选择(复制) 个体选择的方法有很多种(可以自己参照前面的ppt),在我这个例子中,我采用是轮盘赌选择算法
注: 在本例中选择后的种群个体数目和原种群个数相同。
具体实现如selection.py模块
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 import randomdef sum (fit_value ): total = 0 for i in range (len (fit_value)): total += fit_value[i] return total def cumsum (fit_value ): for i in range (len (fit_value) - 2 , -1 , -1 ): t = 0 j = 0 while j <= i: t += fit_value[j] j += 1 fit_value[i] = t fit_value[len (fit_value) - 1 ] = 1 """ @brief 对种群进行选择复制 @param pop 种群,一个二元组 fit_value 适应度列表 @retval 新的种群 """ def selection (pop, fit_value ): newfit_value = [] total_fit = sum (fit_value) for i in range (len (fit_value)): newfit_value.append(fit_value[i] / total_fit) cumsum(newfit_value) ms = [] pop_len = len (pop) for i in range (pop_len): ms.append(random.random()) ms.sort() fitin = 0 newin = 0 newpop = pop while newin < pop_len: if ms[newin] < newfit_value[fitin]: newpop[newin] = pop[fitin] newin = newin + 1 else : fitin = fitin + 1 pop = newpop if __name__ == "__main__" : pop = [[0 , 0 , 1 ], [0 , 0 , 0 ], [0 , 1 , 0 ]] fit = [4 , 4 , 2 ] print (pop) selection(pop, fit) print (pop)
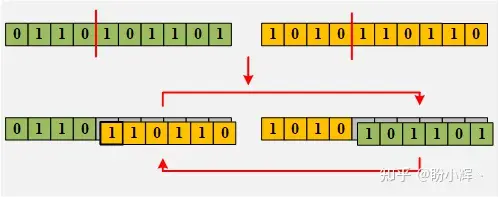
交叉 交叉方法我在这个例子中选择的是单点交叉(Single-point-Crossover) ,单点交叉就是随机选择双亲染色体上的位置。此位置称为交叉点 (crossover point) 或切割点 (cut point)。该点右边的基因在双亲染色体之间交换,得到了两个后代,每个后代都携带着双亲的一些遗传信息。可以配合图像理解
程序实现见crossover.py模块
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 import random""" @brief 采用单点交叉 @param pop 种群,一个二元组 pc 交叉概率 @retval none """ def crossover (pop, pc ): for i in range (len (pop) - 1 ): if random.random() < pc: cpoint = random.randint(0 , len (pop[0 ])) temp1 = [] temp2 = [] temp1.extend(pop[i][0 :cpoint]) temp1.extend(pop[i + 1 ][cpoint : len (pop[i])]) temp2.extend(pop[i + 1 ][0 :cpoint]) temp2.extend(pop[i][cpoint : len (pop[i])]) pop[i] = temp1 pop[i + 1 ] = temp2 if __name__ == "__main__" : pass
变异 变异方式我在本例中采取的是位点变异 ,具体思想就是在染色体的一串基因中,随机找一个位置上的基因进行按位取反(因为采用的编码格式是二进制编码)
程序实现见mutation.py
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 import random""" @brief 采用的变异方法是随机单点变异,意思就是随机在染色体上找一个基因进行按位取反 @param pop 种群,一个二元组 pm 变异概率 @retval none """ def mutation (pop, pm ): for i in range (len (pop)): if random.random() < pm: mpoint = random.randint(0 , len (pop[0 ]) - 1 ) if pop[i][mpoint] == 1 : pop[i][mpoint] = 0 else : pop[i][mpoint] = 1 if __name__ == "__main__" : pass
主函数 介绍完以上七个功能模块就能对主程序进行编写了
具体实现见ga.py
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 import matplotlib.pyplotimport mathfrom calobjValue import calobjValuefrom calfitValue import calfitValuefrom selection import selectionfrom crossover import crossoverfrom mutation import mutationfrom best import bestfrom geneEncoding import geneEncodingprint ("适应度函数为:y = 10 * math.sin(5 * x) + 7 * math.cos(4 * x)" )def b2d (b, max_value, chrom_length ): t = 0 for j in range (len (b)): t += b[j] * (math.pow (2 , j)) t = t * max_value / (math.pow (2 , chrom_length) - 1 ) return t pop_size = 500 max_value = 10 chrom_length = 10 pc = 0.1 pm = 0.01 results = [[]] fit_value = [] fit_mean = [] pop = geneEncoding(pop_size, chrom_length) for i in range (pop_size): obj_value = calobjValue(pop, chrom_length, max_value) fit_value = calfitValue(obj_value) best_individual, best_fit = best(pop, fit_value) results.append([best_fit, b2d(best_individual, max_value, chrom_length)]) selection(pop, fit_value) crossover(pop, pc) mutation(pop, pm) results = results[1 :] results.sort() print (results)print ("y = %f, x = %f\n" % (results[-1 ][0 ], results[-1 ][1 ]))X = [] Y = [] for i in range (pop_size): X.append(i) t = results[i][0 ] Y.append(t) matplotlib.pyplot.plot(X, Y) matplotlib.pyplot.show()
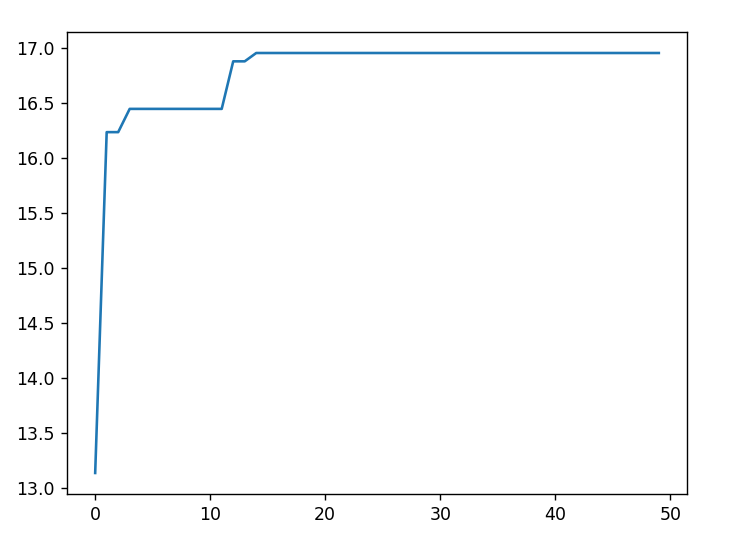
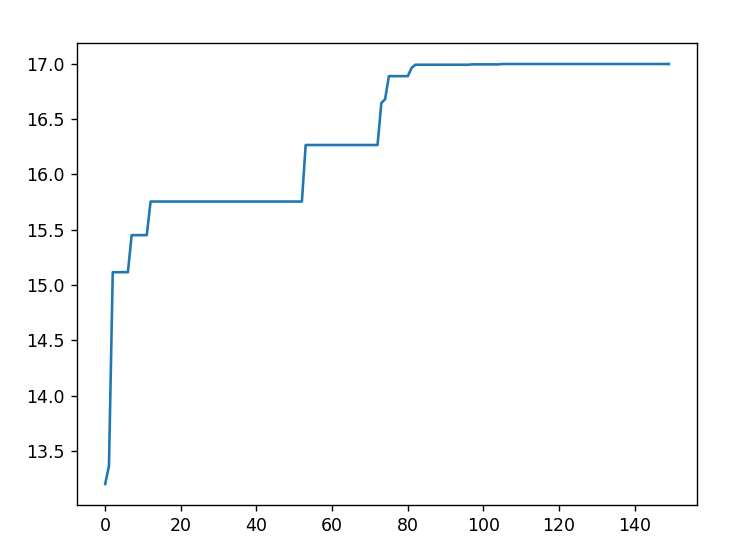
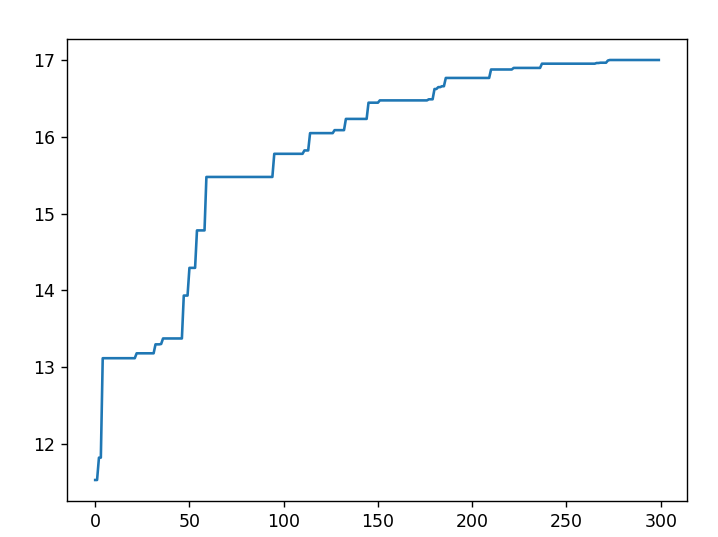
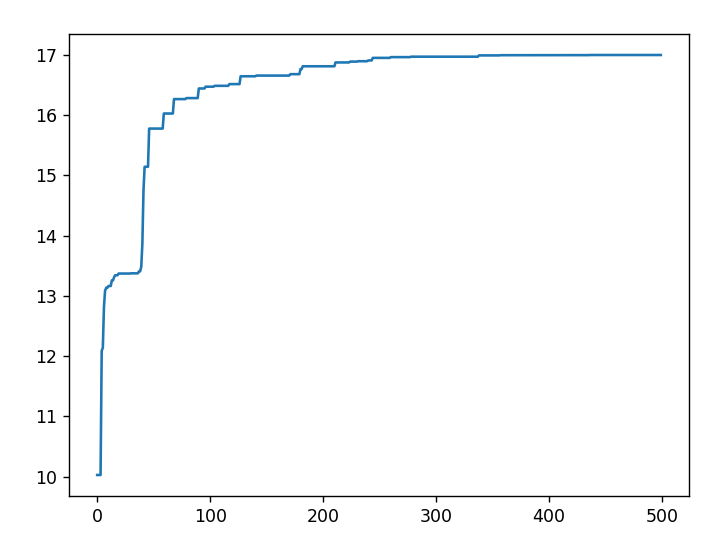
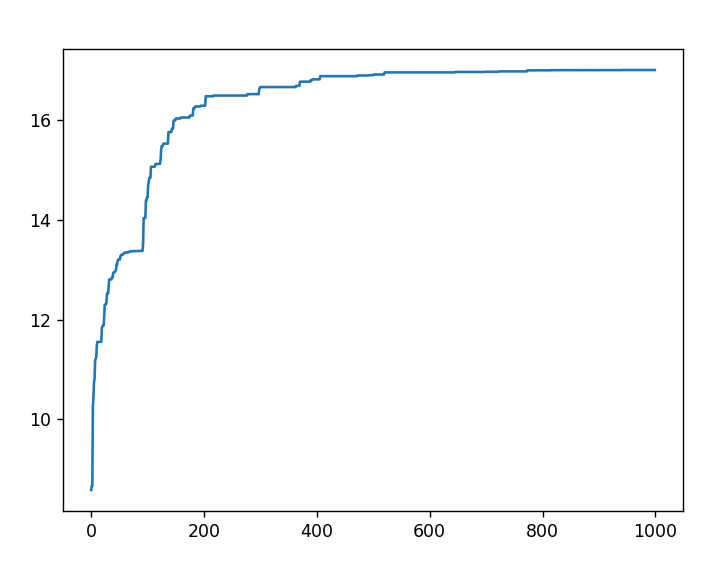
运行结果展示 横坐标为迭代次数,纵坐标为求解结果
迭代次数n=50
迭代次数n=150
迭代次数n=300
迭代次数n=500
迭代次数n=1000
经过多次的结果运行中,我们可以下结论:
函数
的最大值为17 ,同学们也可以自己通过数学运算,验证这一结果
总结与分析 当迭代次数过少的时候,SGA很容易陷入局部最优解 中,但是当迭代次数很多的时候,比如上图测试迭代次数n=1000的结果中,运行时间相当的长(在我自己电脑中有1分钟左右)。所以SGA虽然说操作简单,但是也有非常明显的短板